[Kaggle] Lumbar Spine Degenerative Classification ②
1. 서
저번 포스팅에서 데이터를 이리저리 살펴보며 데이터에 관한 대략적인 그림을 그려놓은 상태이다. 이번에는 파이토치 템플릿을 가져와서 이번 대회에 사용할 수 있도록 커스터마이징을 해보려고 한다. 즉 이번 시간의 목표는 높은 score를 얻는 것이 아닌, 인풋을 받아서 적절한 아웃풋을 내보내는 코드를 완성하고 이를 제출해보는 것이다. 이러한 취지에 따라 일단 모델은 가장 간단하고 바로 만들 수 있는 2층짜리 CNN으로 구성하려고 한다. 물론 score는 낮겠지만 밑바닥부터 시작하여 score를 올려가는 것 또한 즐거운 작업이 될 것이다. 파이토치 템플릿은 이하의 주소에서 가져왔다.
GitHub - victoresque/pytorch-template: PyTorch deep learning projects made easy.
PyTorch deep learning projects made easy. Contribute to victoresque/pytorch-template development by creating an account on GitHub.
github.com
2. 모델 구성 : 베이스
일단 인풋과 관련하여, Spinal Canal Stenosis, Neural Foraminal Narrowing, Subarticular Stenosis 각각에 대하여 모델을 별도로 구성할 필요성이 있다고 판단하였다. 주어진 데이터는 EDA에서 살펴본 바와 같이 Axial T2, Sagatittal T2/STIR, Sagatittal T1 세 종류로 구성되어 있고, T1과 T2 및 Sagittal과 Axial의 특성에 따라 위 세 종류의 데이터들은 각각 Neural Foraminal Narrowing, Subarticular Stenosis, Spinal Canal Stenosis에 적합한 데이터이며, 실제 데이터셋의 라벨링 또한 이에 따라 매칭되어 있기 때문이다.
다음으로 고려할 사항은 아웃풋을 어떻게 해야 할지 여부인데, 이 부분이 당황스러운 부분이었다. 그동안 접해왔던 일반적인 image classification에서 주어진 모든 이미지에 대한 분류 예측 작업을 수행해왔었는데, 본 대회에서 이와 달리 예측에 사용되는 이미지는 주어진 이미지의 일부에 불과했기 때문이다. 여러 방법을 고민하였으나 최종적으로는 모든 이미지에 대해 예측을 수행하고, Severe, Moderate, Mild/Normal 순으로 정렬하여 (사실상 Severe 기준) 가장 높은 값을 갖는 이미지를 기준으로 제출파일을 구성하기로 결정하였다. 분류의 특성상 9개의 이미지에서 Mild 라고 분류했어도 하나의 이미지에서 Severe로 분류했다면 그 환자는 해당 질환이 Severe하다고 판단하는게 옳기 때문이다.
위에서 언급한 기준에 충족하도록 주어진 파이토치 템플릿을 적절히 커스터마이징하였다. 서설에서 언급한 바와 같이 일단은 다음과 같은 가장 간단한 2층짜리 모델을 구성하여 각 모델을 5 epoch씩만 학습을 진행하였다.
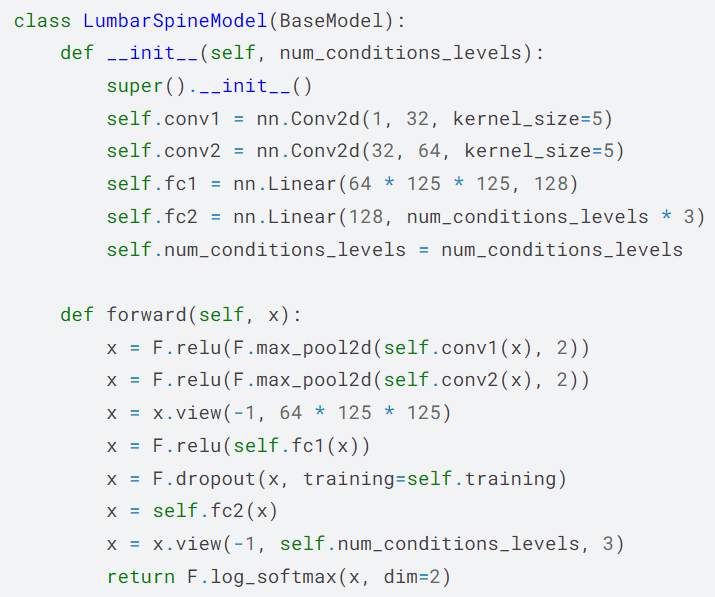
결과물을 제출한 결과 score는 0.93을 기록하여 상위 약 75% 선에 안착하였다. 참고로 본 대회의 평가지표상 score는 낮을 수록 바람직하다.
3. 베이스 모델에서 개선할 것 들
(1) Neural Foraminal Narrowing의 좌우 구별
베이스 모델의 경우 간단한 구조는 논외로 하더라도 한 가지 문제점이 있었다. 다음 데이터를 살펴보자.
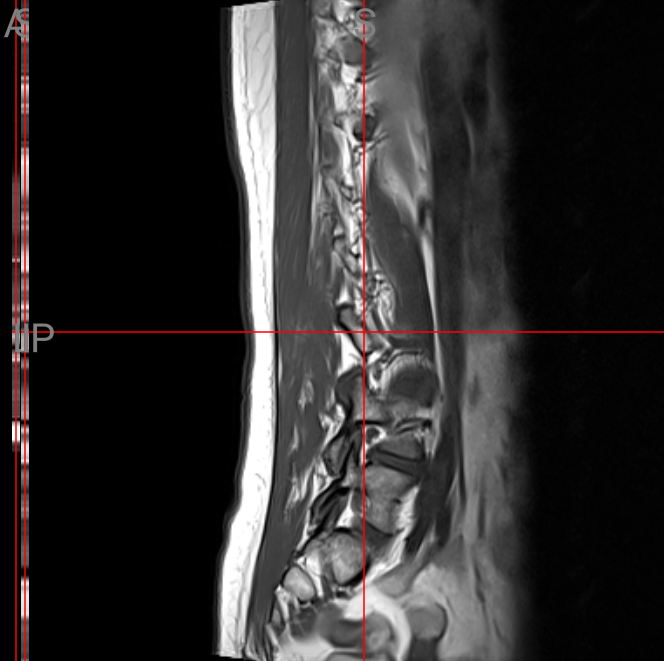
위 데이터는 Sagittal T1, 즉 시상면의 T1 이미지로 Neural Foraminal Narrowing 질환의 중증도 판단에 사용되는 데이터이다. 문제가 되는 것은 Neural Foraminal Narrowing은 Left와 Right를 각각 판단해주어야 하는데, 주어진 데이터의 이미지만으로 모델이 좌우 구별하는 것은 불가능해 보인다는 점이다. Subarticular Stenosis 또한 마찬가지로 Left와 Right를 각각 판단해주어야 하지만, Axial T2인 축방향 T2이미지로 판단하기 때문에 하나의 이미지에서 Left와 Right 모두 판단이 가능하다는 점에서 차이가 있다. 실제로 5 epoch의 학습동안 Average Loss는 Subarticular Stenosis의 경우 0.39, Spinal Canal Stenosis의 경우 0.29까지 감소하였으나 Neural Foraminal Narrowing의 경우 0.57선에 머물러 있었다. 이러한 점을 바탕으로, Neural Foraminal Narrowing의 경우 모델이 좌우 구별을 정확히 할 수 없어 낮은 성능을 보이고 있다고 판단하였다.
따라서 dcm 이미지의 메타데이터를 활용하여, 적어도 테스트셋의 Sagittal T1 데이터는 다시 Left와 Right로 구별한 뒤에 inference를 진행해야 하지 않을까 싶다. 여기에 더하여, train과정에서도 모델도 이에 맞게 따로 구성을 해주어 총 4개의 모델을 학습시키는 것 또한 추가적으로 적용해볼 만하다고 생각한다.
(2) Label이 없는 train 데이터 활용방안
현재 train 데이터셋을 구성하는 dcm 이미지들 중에 Label이 되어있는 것들은 일부에 불과하고, 그 Label은 Normal/Mild, Moderate, Severe 세 가지로 구성되어 있다. 주어진 데이터셋은 인체의 단면을 층마다 찍은 MRI사진으로, 연속성을 갖는다는 특성이 있다. 당연하게도 연속적으로 이루어진 MRI이미지중에서 주어진 여러 질환에 대한 예측이 가능한 데이터셋은 일부에 불과하다. 질환이 발생하는 부위를 담고 있는 사진은 일부에 불과할 것이기 때문이다.
test 데이터셋 또한 마찬가지의 구성을 갖고 있다. 결국 모델이 예측을 수행할 경우에는 예측이 가능한 데이터와 함께 예측을 할 수 없는 데이터도 함께 참고하게 될 것이다. 이를 위 세 가지 label로 분류하는 것은 모델의 성능하락의 원인 중 하나가 될 수 있을 것이다. 따라서, 레이블 되어 있지 않은 데이터셋에 대하여는 별도의 label을 지정하여 네 가지 label로 분류하는 모델로 재구성하면 성능을 조금은 더 올릴 수 있지 않을까 생각된다.
4. 결어
개선할 사항이 아직 여럿 있지만, 캐글 대회에서 스스로의 힘으로 모델을 구성하여 제출까지 완료했다는 점은 굉장히 뿌듯한 부분이다. 다음 포스팅에서 위 개선사항과 함께 성능이 좋은 모델을 가져와 score를 최대한 끌어올리며 캐글 대회를 마무리해보려고 한다.